【alvaDesc】機能に関するFAQ
alvaDesc TOPはこちら
alvaDesc よくある質問はこちら
目次
記述子を保存する際の変数削減(Variable reduction)オプションの詳細
A. 「Save descriptors」→「Variable reduction」で出力する記述子の数を減らす変数削減として6つのオプションがあります。
このオプションを利用することで、その後の解析の際に邪魔になる記述子を取り除き、データサイズを小さくすることができます。
- Constant values:
すべての値が同じ場合(定数)、その記述子は保存されません。
- Near constant values:
記述子が、1つの値だけが異なり残りの値が全て同じ場合(定数)、“near constant value”とみなされその記述子は保存されません。
- Standard deviation less than:
標準偏差が指定された閾値より小さい記述子は保存されません。
- At least one missing value:
一つでも欠損値(na)がある記述子は保存されません。
- All missing values:
すべてが欠損値(na)となっている記述子は保存されません。
- Pair correlation larger or equal to:
高速V-WSPアルゴリズム※を用いて、指定された閾値以上の相関係数を示す記述子ペアのどちらか一方を自動的に削除します。尚、閾値は相関係数の絶対値を表わしており、正の相関、負の相関共に削除が行われます。
※V-WSPアルゴリズム:
記述子の削減は以下の手順で行われます。
ⅰ.最も相関係数の値が高い記述子のペアを抽出。
ⅱ.抽出された二つの記述子から他の記述子との相関係数の総和を算出して比較し、値が高い方(似通った記述子がより多い方)の記述子を取り除く。
ⅲ.指定された閾値以上の相関係数を持つ記述子ペアが他に存在しているのであれば、ⅰ.に戻り作業を繰り返す。
参考文献
Ballabio, D., Consonni, V., Mauri, A., Claeys-Bruno, M., Sergent, M., & Todeschini, R. (2014), A novel variable reduction method adapted from space-filling designs, Chemometrics and Intelligent Laboratory Systems, 136, 147–154.
(2021/5/27) (2022/6/9追記)
非完全結合型の分子に対して記述子を計算させる方法
A. 塩、混合物、イオン液体、金属錯体など、非完全結合型の分子構造(disconnected structures)に対しても記述子が計算できることがalvaDescの特徴の一つです。
入力方法【SMILES】
SMILES表記法を使ってalvaDescに入力する場合、それぞれの個別構造が”.”で区切られたSMILESに於ける非完全結合型分子表現を用いることで入力します。
- 塩の例
- テトラクロロ亜鉛酸アンモニウム(Cl4H8N2Zn, CAS 14639-97-5)
[NH4+].[NH4+].[Cl-].[Cl-].[Cl-].[Cl-].[Zn+2]

- 混合物の例
- トルエン(C7H8, CAS 108-88-3)とベンゼン(C6H6, CAS 71-43-2)の混合物
CC1=CC=CC=C1.C1=CC=CC=C1
- トルエンとベンゼンが2:1の混合物
CC1=CC=CC=C1.CC1=CC=CC=C1.C1=CC=CC=C1
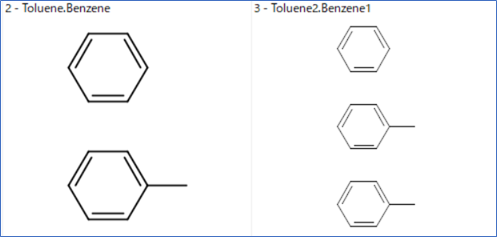
- イオン液体の例
- 硝酸エチルアンモニウム(C2H8N2O3, CAS 22113-86-6)
CCN.[N+](=O)(O)[O-]

- 金属錯体の例
- フェロセン(C10H10Fe, CAS 102-54-5)
[CH-]1C=CC=C1.[CH-]1C=CC=C1.[Fe+2]

入力方法【SDFフォーマット、MOL2フォーマット】
非完全結合型の分子をSDFフォーマットやMOL2フォーマットなど、結合テーブルを持ったフォーマットで入力する場合、それぞれの個別構造間に結合がないため、結合テーブルは個別構造に対応した結合テーブルを順に並べたような形となります。
- 塩の例
- テトラクロロ亜鉛酸アンモニウム(Cl4H8N2Zn)(2D-SDFフォーマット)

2個の[NH4+]、4個の[Cl-]と[Zn+2]がありますが、それぞれに結合がないので、結合テーブルはありません。原子テーブルでは左から6番目の形式電荷を表わす場所には、Nが3(+1の形式電荷)、Clが5(-1の形式電荷)、Znが2(+2の形式電荷)と表現されています。また、最後のプロパティーライン(M CHG)に、対象となる7つの原子それぞれの番号と対応する形式電荷が表示されています。
- 混合物の例
- トルエン(C7H8)とベンゼン(C6H6)の混合物(3D-SDFファイル)

原子テーブルには各原子の3次元座標がX/Y/Zで表示されています。結合テーブルでは、ベンゼンに関する#1のCから#12のHに対する結合テーブルに、トルエンに関する#13のCから#27のHに対する結合テーブルが付け加えられた形となっています。
- トルエン(C7H8)とベンゼン(C6H6)が2:1の混合物(2D-SDFフォーマット)

2D-SDFフォーマットですので、原子テーブルはXとYの値のみでZの値は全て0となっています。結合テーブルでは、最初のトルエンに関する#1から#7のCに対して定義された結合テーブルと、2番目のトルエンに関する#8から#14のCに対する結合テーブルと、ベンゼンに関する#15から#20のCに対する結合テーブルの3つが足し合わされています。
- イオン液体の例
- 硝酸エチルアンモニウム(C2H8N2O3)(2D-SDFフォーマット)

[CCN]の構造に関する#1のCから#3のNの結合テーブルに、[N+](=O)(O)[O-]の構造に関する#4のNから#7のOの結合テーブルが足され、#4のNと#7のOに+1と-1の形式電荷が付加されています。
- 金属錯体の例
- フェロセン(C10H10Fe)(3次元座標と原子の実効電荷を持つmol2フォーマット)

他の例と同様に、結合テーブルでは、C1からH10までの結合とC12からH21までの結合のみが定義され、Fe11は結合テーブル上にはありません。また、Fe11の形式電荷+2は原子テーブル上の実効電荷1.622069で表現されています。
入力した3次元座標を用いて分子の画像を表示させると、Z軸方向の上から俯瞰した2次元画像となります。

計算オプション
alvaDescでは、非完全結合型分子に対して記述子を計算する際に6つの計算アプローチを使用することができます。
- 非完全結合型の分子全体を1つの分子として扱い、全体を記述子の定義に基づき計算するアプローチ《スタンダード》(デフォルト)
- 結合していない構造それぞれを1つの分子として扱い、それぞれに対して記述子を計算するアプローチ
- 最大値を記述子計算の結果とする《最大値》
- 最小値を記述子計算の結果とする《最小値》
- 平均値を記述子計算の結果とする《平均値》
- 合計値を記述子計算の結果とする《合計値》
- 結合していない構造の1つだけを分子として扱うアプローチ
- 水素数が一番多い完全結合型の最大構造のみに対して記述子を計算し、非完全結合型分子全体の値とする 《最大フラグメントのみを保持》
デフォルト設定である《スタンダード》アプローチでは計算できないものも出てきますが、その場合でも、他のアプローチに変更することで記述子が計算できるようになります。
(2023/7/20)
全記述子のデータタイプ(整数、浮動小数点数、ブーリアンなど)がまとめられた表
ECFP4を計算させるための設定
A. alvaDescのフィンガープリントの計算オプションでは、ECFPの場合、中心原子からの最大半径をMaximum lengthと表示しています。ECFP4などフィンガープリント名の最後につく数字は直径を表しておりますので、Maximum length = 半径(radius) = 2 の場合、直径(diameter)は4となります。ビット長は一般的に1024ビットが使用されていますので、alvaDescのECFP計算をデフォルト設定で行いますと、ECFP4を計算させていることになります。
また同様に、PFP計算ではMaximum lengthは最大のパス長(maximum path length)を表しています。
尚、計算されるフィンガープリントはハッシュ化計算がされておりますので、他のツールやソフトウェアから得られた同様のフィンガープリント(例えばECFP4)と同列に比較することはできません。
(2021/5/25)
Editメニューの “Copy as SMILES”で生成されるSMILES(Canonical SMILESか)
A. “Copy as SMILES”で生成されるSMILESはCanonical SMILESです。
alvaDescは、入力された分子に対してニトロ基の標準化と芳香族化の標準化を行った後でcanonicalize(正準化)してSMILESに変換します。
但し,canonicalizeのアルゴリズムはツール毎に違っており、alvaDescで生成したものが他のツールで生成したSMILESと異なるケースもあり得ます。alvaDescの場合立体化学を考慮していないため、同じ立体化学をもつ同じ分子でも異なるSMILESになることもあり得ます。
次の文献をご参照ください。
Neglur, G., Grossman, R. L., & Liu, B. (2005). Assigning Unique Keys to Chemical Compounds for Data Integration: Some Interesting Counter Examples. In Data Integration in the Life Sciences (Vol. 3615, pp. 145–157). https://doi.org/10.1007/11530084_13)
(2022/10/4)
ユーザーマニュアルの参照
A. ユーザーマニュアルは、下記の方法によりご参照ください。
alvaDescの起動後、画面上部のメニューより、「Help」を選択し、「alvaDesc help」をクリックします。
Webブラウザが起動され、「alvaDesc x.x – User manual」が表示されます。
(2019/05/15) (2023/08/01修正)
コマンドラインインターフェース(CLI)からの使用方法
A. CLIからの使用は、入力する化合物がとても多い場合や他のプログラムと連携して使用する場合など、alvaDescの計算をバックグラウンドでバッチ処理できるので便利です。alvaDescをCLIから使用するには、alvaDescのGUI実行プログラムと同時にインストールされるalvaDescCLI実行ファイルを使用します。
デフォルト格納ディレクトリ
| Windows : | C:/Program Files/Alvascience/alvaDesc/alvaDescCLI.exe |
| Linux : | /usr/bin/alvaDescCLI |
| MacOS : | /Applications/alvaDesc.app/Contents/MacOS/alvaDescCLI |
以下の使用例では、環境変数PATH/Pathにこれらのディレクトリが登録されており、alvaDescCLI実行ファイルを(パス指定することなく)直接実行できることを前提としています。
使用例(Windows/Linux/MacOS共通)
1つのSMILES文字列を入力として,指定記述子を計算
alvaDescCLI --iSMILES=CO --descriptors=MW,MLOGP出力
alvaDescCLI.
Version 2.0 for Win64 (x86_64) - v.2.0.14 - built on: 2022/11/11
alvaDesc Commercial Site license. Expiration date: 2023/12/31
Licensee: Affinity Science Corporation
Date: 木曜日, 09 2月 2023
Time: 16:51:40
32.05 -0.814MACCS166計算
alvaDescCLI --iSMILES=CCO --maccsfp標準入出力を使用した記述子計算
alvaDescCLI --input --inputtype=SMILES --descriptors=MW,nAA,ChiralCenter,MLOGP --labels < sample.smi > output.txt- iSMILESの場合は、descriptorsで指定した記述子の順序が考慮されますが、それ以外では、記述子リストの順番で出力されます。labelsオプションでご確認ください。
input/outputオプションを使用した入出力ファイルの指定
alvaDescCLI --descriptors=ALL --input=sample.sdf --outpupt=output.txt --labels- 入力ファイルの拡張子でファイル形式が判定されます。
コマンドヘルプの表示
alvaDescCLI -hパイプを利用した入力構造の指定
echo CCO | alvaDescCLI --input --inputtype=SMILES --ecfp- Linux/MacOSでは、”echo -e “C\nCC\nCCC”などとして、改行コードを含めることで複数化合物の一括処理も可能です。
パイプとファイルを利用した入力構造の指定
(Windows)
type sample.smi | alvaDescCLI --input --inputtype=SMILES --descritpors=ALL2D(Linux/MacOS)
cat sample.smi | alvaDescCLI --input --inputtype=SMILES --descritpors=ALL2DalvaDescCLIの詳細は、GUIのHelp メニューから alvaDesc Help を選択し、表示されるマニュアルの3. Command-line Interfaceをご参照ください。
(2023/2/9)
Pythonからの使用方法
A. alvaDescCLIWrapperをインストールすることにより、PythonからalvaDescCLIを呼び出して使えるようになります。次の手順に従ってインストールを行い、サンプルプログラム例を参照してお使いください。
インストール手順
1. ダウンロードページ(alvaDesc本体と同じ弊社DLページ)よりZIPファイルをダウンロードし、任意のディレクトリに展開します。
2. alvaDescCLIWrapperフォルダーの中に入っているセットアッププログラムをコマンドプロンプト上で実行します。
python setup.py install実行要件
・ライセンスが付与されたバージョン 1.0.14以上のalvaDescがインストールされていること。
・Python 3.5以上がインストールされていること。
サンプルプログラム(Windows)
SMILES表記されたブタンとアスピリンのmolecular weight (MW)とaverage molecular weight (AMW)を計算させます。
from alvadesccliwrapper.alvadesc import AlvaDesc
# aDesc = AlvaDesc("C:/Program Files/Alvascience/alvaDesc/alvaDescCLI.exe")
# To remove blank space, use the command "dir /x" to confirm the short name.
# For example, > cd c:\ & dir /x
aDesc = AlvaDesc("C:/Progra~1/Alvascience/alvaDesc/alvaDescCLI.exe")
aDesc.set_input_SMILES(['CCCC', 'CC(=O)OC1=CC=CC=C1C(=O)O']) # set the molecules SMILES
if not aDesc.calculate_descriptors(['MW', 'AMW']):
print('Error: ' + str(aDesc.get_error()))
else:
print('Results: ' + str(aDesc.get_output()))実行結果

※実行結果の見方
| Molecule | MW | AMW |
|---|---|---|
| CCCC | 58.14 | 4.1529 |
| CC(=O)OC1=CC=CC=C1C(=O)O | 180.17 | 8.5795 |
alvaDescCLI実行プログラムのデフォルト格納場所
- Windows(*)
- C:/Program Files/Alvascience/alvaDesc/alvaDescCLI.exe
- MacOS
- /Applications/alvaDesc.app/Contents/MacOS/alvaDescCLI
- Linux
- /usr/bin/alvaDescCLI
(*)”Program Files”に含まれるスペースが問題になるため,上記サンプルプログラムのようにMS-DOS短縮名に置き換える必要があります.このような場合,コマンドプロンプト等で親ディレクトリへ移動し,”dir /x”コマンドを実行し,短縮後のフォルダ名を確認することができます.下図で黄色で囲んだ”PROGRA~1″が短縮名になります.

入出力ファイルの指定
- MDLファイルからの入力
- aDesc.set_input_file(‘ファイル名.sdf’, ‘MDL’)
- テキストファイルへの出力
- aDesc.set_output_file(‘ファイル名.txt’)
- set_output_fileを使用すると計算結果はalvaDescの標準形式でファイルに出力されます。get_outputを使用する場合はこの機能を使用することができません。
記述子の計算
- 選択した記述子の計算
- aDesc.calculate_descriptors([‘記述子1’, ‘記述子2’, …])
- 全ての記述子の計算
- aDesc.calculate_descriptors(‘ALL’)
フィンガープリントの計算
- MACCS166
- aDesc.calculate_fingerprint(‘MACCSFP’)
- ECFP(1024ビット長)
- aDesc.calculate_fingerprint(‘ECFP’, 1024)
NumPy/Pandas dataframeへの記述子計算結果のコンバート
get_output、get_output_descriptors、get_output_molecule_namesを使用してNumPyやPandas dataframeに計算結果のデータをコンバートすることができます。(alvaDescCLIWrapper ver. 1.0.4より)
サンプルプログラム
import numpy as np
import pandas as pd
from alvadesccliwrapper.alvadesc import AlvaDesc
aDesc = AlvaDesc() # Windows is the default
aDesc.set_input_SMILES([‘C#N’, ‘CCCC’, ‘CC(=O)OC1=CC=CC=C1C(=O)O’])
if not aDesc.calculate_descriptors([‘AMW’, ‘MW’, ‘nBT’]):
print(‘Error: ‘ + aDesc.get_error())
else:
res_out = aDesc.get_output()
# get molecule names according to alvaDescCLI standard
res_mol_names = aDesc.get_output_molecule_names()
res_desc_names = aDesc.get_output_descriptors()
# NumPy array of array and matrix
numpy_array_of_array = np.array([np.array(xs) for xs in res_out])
numpy_matrix = np.matrix(res_out) # NumPy matrix
print(‘NumPy matrix’)
print(numpy_matrix)
# Pandas dataframe
pandas_df = pd.DataFrame(res_out)
pandas_df.columns = res_desc_names
pandas_df.insert(loc=0, column=’NAME’, value=res_mol_names)
print(‘Pandas dataframe’)
print(pandas_df)結果
NumPy matrix
[[ 4.1529 58.14 13. ]
[ 8.5795 180.17 21. ]]
Pandas dataframe
NAME AMW MW nBT
0 Molecule1 4.1529 58.14 13.0
1 Molecule2 8.5795 180.17 21.0使用可能なクラス・メソッド
alvaDescCLIWrapperで使用可能なクラス・メソッドの説明ドキュメントは、zipファイル展開後、docフォルダ内のHTMLファイルをご参照ください(例. v1.0.4の alvadesc.html)。
(2021/7/8)
Ubuntu環境でのalvaDescCLIWrapperのセットアップと使用方法
A. PythonでalvaDescCLIWrapperをセットアップすることにより、Ubuntu環境に既にインストールされているalvaDescをPythonから呼び出して使用することができます。なお、alvaDescは1.0.14以上、Pythonは3.5以上が必要です。
・準備
alvaDescCLIのインストールディレクトリとライセンス状況を確認します。
egghead@DESKTOP-BKJHQMR:~/temp$ which alvaDescCLI
/usr/bin/alvaDescCLI
egghead@DESKTOP-BKJHQMR:~/temp$ alvaDescCLI --license
Your license will expire on 2023/3/31
alvaDescCLI.
Version 2.0 for Linux (x86_64) - v.2.0.14 - built on: 2022/11/11
Your license is valid
License file: /home/egghead/.config/Alvascience/alvaDesc/alvalicense_alvaDesc.alic
Organisation: Affinity Science Corporation
Contact: Affinity Science Corp.
Use: Commercial
Type: Site
Expiration: 2023/3/31
egghead@DESKTOP-BKJHQMR:~/temp$※Linux用のデフォルトでは、alvaDescCLI実行プログラムは /usr/bin/alvaDescCLI に格納されています。デフォルトディレクトリ以外に格納されていましたら、適宜変更ください。
・セットアップ
まず、ダウンロードしたalvaDescCLIWrapperのzipファイルを展開します。
egghead@DESKTOP-BKJHQMR:~/temp$ unzip python-alvaDesc_all_0_v1_0_6.zip
Archive: python-alvaDesc_all_0_v1_0_6.zip
creating: alvaDescCLIWrapper/
creating: alvaDescCLIWrapper/alvadesccliwrapper/
inflating: alvaDescCLIWrapper/alvadesccliwrapper/alvadesc.py
extracting: alvaDescCLIWrapper/alvadesccliwrapper/__init__.py
inflating: alvaDescCLIWrapper/CHANGES.txt
creating: alvaDescCLIWrapper/doc/
inflating: alvaDescCLIWrapper/doc/alvadesc.html
inflating: alvaDescCLIWrapper/LICENSE.txt
inflating: alvaDescCLIWrapper/README.txt
inflating: alvaDescCLIWrapper/setup.py
egghead@DESKTOP-BKJHQMR:~/temp$次に、セットアップスクリプトを実行します。
egghead@DESKTOP-BKJHQMR:~/temp$ cd alvaDescCLIWrapper/
egghead@DESKTOP-BKJHQMR:~/temp/alvaDescCLIWrapper$ sudo python3 setup.py install
running install
(途中省略)
Installed /usr/local/lib/python3.8/dist-packages/alvadesccliwrapper-1.0.0-py3.8.egg
Processing dependencies for alvadesccliwrapper==1.0.0
Finished processing dependencies for alvadesccliwrapper==1.0.0
egghead@DESKTOP-BKJHQMR:~/temp/alvaDescCLIWrapper$ cd ..
egghead@DESKTOP-BKJHQMR:~/temp$
※この例ではシステムディレクトリにインストールするため管理者権限が必要です。
・サンプルスクリプトの作成と実行
セットアップが終了したら、次のサンプルプログラムscript.pyを作成してみます。
from alvadesccliwrapper.alvadesc import AlvaDesc
def get_descriptors_from_block(list_descriptors, block_name):
# [1]: Name
# [3]: Block
descs = [desc[1] for desc in list_descriptors if desc[3] == block_name]
return descs
aDesc = AlvaDesc('/usr/bin/alvaDescCLI')
list_descriptors = aDesc.get_descriptors()
const_descriptors = get_descriptors_from_block(list_descriptors, 'Constitutional indices')
aDesc.set_input_SMILES(['C#N', 'CCCC', 'CC(=O)OC1=CC=CC=C1C(=O)O'])
if not aDesc.calculate_descriptors(const_descriptors):
print('Error: ' + aDesc.get_error())
else:
print(aDesc.get_output_descriptors())
print(aDesc.get_output())
このサンプルは、SMILESで与えられた3つの分子’C#N’、‘CCCC’、‘CC(=O)OC1=CC=CC=C1C(=O)O’に対して’Constitutional indices’のブロックに含まれる記述子を計算させるプログラムです。alvaDescCLIのパスはwhichコマンドの出力等を参照し、適宜修正して下さい。
次に、作成したサンプルスクリプトを実行します(sample.pyはtempディレクトリに保存されている前提です)。
egghead@DESKTOP-BKJHQMR:~/temp$ python3 script.py
['MW', 'AMW', 'Sv', 'Se', 'Sp', 'Si', 'Mv', 'Me', 'Mp', 'Mi', 'GD', 'nAT', 'nSK', 'nAA', 'nTA', 'nBT', 'nBO', 'nBM', 'SCBO', 'RBN', 'RBF', 'nDB', 'nTB', 'nAB', 'nH', 'nC', 'nN', 'nO', 'nP', 'nS', 'nF', 'nCL', 'nBR', 'nI', 'nB', 'nHM', 'nHet', 'nX', 'H%', 'C%', 'N%', 'O%', 'X%', 'nCsp3', 'nCsp2', 'nCsp', 'Fsp3', 'max_conj_path', 'nStructures', 'totalcharge'] [[27.03, 9.01, 2.0214, 3.1018, 2.0057, 3.4983, 0.6738, 1.03393333333333, 0.668566666666667, 1.1661, 1.0, 3.0, 2.0, 0.0, 2.0, 2.0, 1.0, 1.0, 3.0, 0.0, 0.0, 0.0, 1.0, 0.0, 1.0, 1.0, 1.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 1.0, 0.0, 33.3333333333333, 33.3333333333333, 33.3333333333333, 0.0, 0.0, 0.0, 0.0, 1.0, 0.0, 0.0, 1.0, 0.0], [58.14, 4.15285714285714, 6.634, 13.418, 7.807, 16.076, 0.473857142857143, 0.958428571428572, 0.557642857142857, 1.14828571428571, 0.5, 14.0, 4.0, 0.0, 2.0, 13.0, 3.0, 0.0, 3.0, 1.0, 0.0769230769230769, 0.0, 0.0, 0.0, 10.0, 4.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 71.4285714285714, 28.5714285714286, 0.0, 0.0, 0.0, 4.0, 0.0, 0.0, 1.0, 0.0, 1.0, 0.0], [180.17, 8.57952380952381, 13.9664, 21.8436, 13.8636, 23.4984, 0.665066666666667, 1.04017142857143, 0.660171428571428, 1.11897142857143, 0.166666666666667, 21.0, 13.0, 6.0, 4.0, 21.0, 13.0, 8.0, 18.0, 3.0, 0.142857142857143, 2.0, 0.0, 6.0, 8.0, 9.0, 0.0, 4.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 0.0, 4.0, 0.0, 38.0952380952381, 42.8571428571429, 0.0, 19.047619047619, 0.0, 1.0, 8.0, 0.0, 0.111111111111111, 8.0, 1.0, 0.0]]
egghead@DESKTOP-BKJHQMR:~/temp$この例では、記述子記号、値が標準出力に表示されています。
・使用可能なクラス・メソッド
alvaDescCLIWrapperで使用可能なクラス・メソッドの説明ドキュメントは、zipファイル展開後にdocフォルダ内のHTMLファイルをご参照ください(alvaDescCLIWrapper/doc/alvadesc.html)。簡単なサンプルプログラムも掲載されています。
(2023/1/19)
alvaDesc Knimeプラグインのインストール方法
A. alvaDesc プラグインをKNIMEから直接ダウンロード可能です。
- まず,KNIMEバージョンが、3.7.1以降であることを確認してください。
(*これより古いバージョンの場合、KNIME自体のアップデートをご検討ください) - KNIME操作画面から、File > Preference > Install/update > Available Software Sitesとして、「Alvascience update site」が存在するか確認してください.存在しない場合は,次の操作を行いサイトロケーションを追加してください。
- File > Preferences > Install/Update > Available Software Sitesを選択
- Add…ボタンをクリック
- Name欄に「Alvascience update site」を入力
- Location欄に「https://www.alvascience.com/knime/latest」を入力
- Addボタンをクリック
- Apply and Closeボタンをクリックして終了
- 続けて,File > Install KNIME Extensions…を選択します。
- リストの中から「Alvascience ~」を探し、「Alvascience alvaDesc Plugin」を選択します。
- Nextボタンをクリックし、以降はKNIMEの指示に従い、インストールを進めてください.
(2020/09/23)
