LigandScout
ロバストなアライメントアルゴリズムとユーザフレンドリなUIをもつファーマコフォアのモデリング・スクリーニング解析ソフトウェア
目次
製品概要
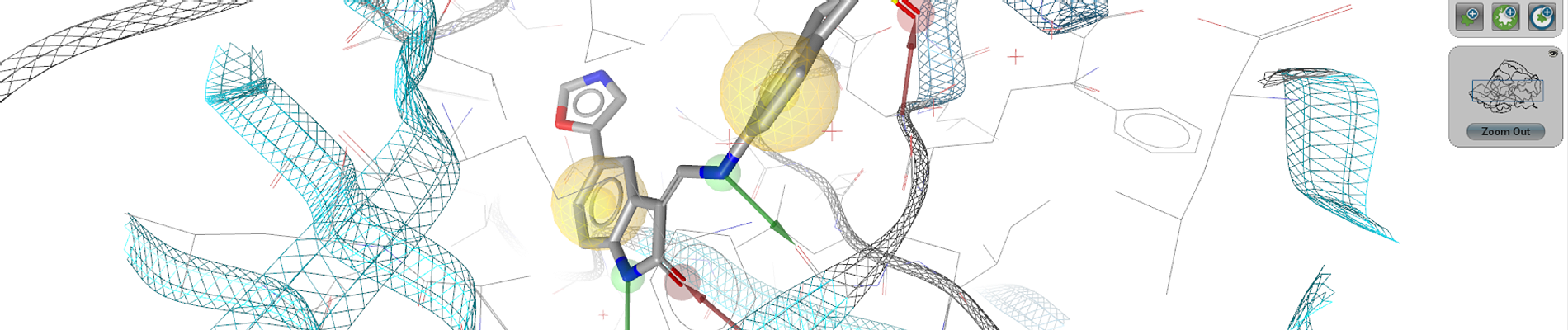
LigandScout は、ファーマコフォア(薬理作用団)モデルに基づく、インシリコ創薬のための統合ソフトウェアです。独自のアライメントアルゴリズムにより、構造ベースとリガンドベースのファーマコフォアモデルの効果的な作成、高速・高精度なインシリコスクリーニング等が可能です。
また、アポタンパク・ファーマコフォアモデリング、結合ポケット探索、低分子ドッキング、分子動力学計算との連携、クラウド・コンピューティングなど,当該分野の最新手法が組み込まれており、活発な開発が続けられています。
ワークフローに最適化されたGUIに加え、各種コマンドラインツール、KNIME向け拡張ノード等が実装されており、目的に応じて様々な利用形態をお選びいただけます。
開発元:オーストリア inte:Ligand GmbH
主要な機能
構造ベースファーマコフォア・モデリング
- 複合体構造からリガンド周辺構造を考慮したファーマコフォアモデルの自動生成
- ジオメトリ、辞書およびルールに基づくリガンド自動認識
- 動的に連動した3D/2D分子ビューと階層ビュー、化合物ライブラリビュー
- タンパク-リガンド相互作用の2D表示機能(構造式表示)
- 共通ファーマコフォアの作成(Merged/Shared)
- 補因子、イオン、水分子、金属結合(Fe, Mg, Zn, Mn)の考慮
- ドッキング機能(Autodock/Vinaビルトイン)
- 結合自由エネルギー予測
- リガンド構造調整(簡易分子モデリング機能)
- 水分子および補因子をリガンド、または高分子の一部として扱うことが可能
- MMFF94力場による構造最適化
- ファーマコフォア生成パラメータの調整機能(上級ユーザ向け)
- 多数の分子ファイル形式(SMILES, SDF, MOL2, SMI etc.)と画像ファイル形式(EPS, PDF, JPG, PNG etc.)をサポート
- 互変異性体の生成
- アポタンパクモデリング
- MDトラジェクトリファイルの読み込みと解析、動的ファーマコフォアの生成
リガンドベースファーマコフォア・モデリング
- 活性化合物の構造情報からファーマコフォアモデルを生成
- ファーマコフォアに基づく化合物の分類(クラスタリング)
- 配座発生プログラムiCon、配座解析
- 分子アライメント
- スコア関数(Pharmacophore-Fit, Atom sphere overlap, Gaussian shape similarity etc.)
- 排除体積の自動生成
- 分子記述子計算(MW, cLogP, TPSA etc.)
- SMARTSパターンによるユーザー定義要素
- 類似化合物検索(Online PDB Lookup)
スクリーニング
- ファーマコフォアクエリによる高速・高精度スクリーニング
- モデル評価のためのROC曲線や濃縮係数(EF値)の算出
- パラレルスクリーニング機能(ブール式によるクエリ条件設定)
- スクリーニングモード(Match all query features, Fragment screening)
KNIME拡張
- LigandScoutの多数の機能をKNIME環境(www.knime.org)で利用可能
- リガンド・プロファイリング
- 毒性予測
技術情報
機能比較
Essential、Advanced、Expert、KNIME Extensionsの機能比較表
補足事項:
*Expertには、KNIME Extensionsライセンスが含まれます。
*KNIME Extensionsライセンスには、ILファーマコフォアデータベース(有償オプション)を追加可能です。
| 利用可能な機能 | Essential | Advanced | Expert | KNIME Extensions |
|---|---|---|---|---|
| 構造ベースファーマコフォア・モデリング | ● | ● | ● | ● |
| リガンドベースファーマコフォア・モデリング | ● | ● | ● | ● |
| バーチャルスクリーニング | ● | ● | ● | ● |
| 配座生成(iCon) | ● | ● | ● | ● |
| 互変異生体生成 | ● | ● | ● | ● |
| ファーマコフォアベース・クラスタリング | ● | ● | ● | ● |
| モデルバリデーション機能(ROC描画) | ● | ● | ● | ● |
| 2D分子エディター | ● | ● | ● | |
| インタラクティブグラフ機能 | ● | ● | ||
| 結合ポケット探索 | ● | ● | ||
| 低分子ドッキング(Autodock/Vina) | ● | ● | ● | |
| リガンド結合エネルギー計算 | ● | ● | ||
| アポサイト・ファーマコフォア生成 | ● | ● | ||
| MDトラジェクトリ・インポート | ● | ● | ● | |
| MDトラジェクトリ・ファーマコフォア解析 | ● | ● | ||
| MDトラジェクトリ・ファーマコフォアグラフ機能 | ● | |||
| リモート計算(HPCクラスタ/Amazonクラウド) | ● | ● |
リファレンス
- Wolber, G.; Dornhofer, A. A.; Langer, T.; Efficient overlay of small organic molecules using 3D pharmacophores J. Comput. Aided Mol. Des.; 2007; 20(12); 773-788. DOI: 10.1007/s10822-006-9078-7
- Wolber, G.; Langer, T.; LigandScout: 3-D Pharmacophores Derived from Protein-Bound Ligands and Their Use as Virtual Screening Filters. J. Chem. Inf. Model; 2005; 45(1); 160-169. DOI: 10.1021/ci049885e
- Kainrad T., Hunold S., Seidel T., Langer T.; LigandScout Remote: A New User-Friendly Interface for HPC and Cloud Resources
J. Chem. Inf. Model; 2018. DOI: 10.1021/acs.jcim.8b00716 - Wolber G., Dornhofer A. A., Langer T.; Efficient Overlay of Molecular 3D Pharmacophores. Oral presentation at the spring ACS meeting in Atlanta, GA, USA, 2006 Presentation as PDF file[8.8 MB]
- Wolber, G.; Kirchmair H.; Langer, T.; Structure-Based 3D Pharmacophores: An Alternative to Docking? Plenary talk at the 7th International Conference on Chemical Structures in Noordwijkerhout, 2005 Presentation as PDF file[2.8 MB]
バージョン情報
バージョン情報はこちら
動作環境
対応OS
- 64-bit Linux
- 64-bit Windows
- 64-bit macOS
システム要件
- Windows/Linux
- intel core i5 同等以上の性能を有するIntel64またはAMD64プロセッサ
- macOS
- Intel/ARMベースプロセッサ
- CPUコア毎2GB超の物理メモリ(8GB以上のRAMを推奨)
- ハードウェアアクセラレータを利用可能な3Dグラフィックカード(NVIDIA製カード推奨)
OSバージョン、グラフィックカード、ドライバ等の組み合わせによっては、画面表示が乱れる等の異常動作が生じる場合があります。評価ライセンスやデモモード等により、事前に動作状況を確認されることをお勧めします。
ライセンス形態
LigandScoutのライセンスは、所属機関(一般・官公庁・教育機関)とライセンス(1-seat/Group/Site)の形態、利用可能な機能によって異なります。
ライセンス数による分類
一般用ライセンスには、1-seat/Group/Siteの3つの形態、官公庁用・教育機関用ライセンスには、1-seat/Siteの2つの形態があります。
- 1-seatライセンス
- 1台のコンピュータのみにインストールできます。
- Groupライセンス
- 複数のライセンスをグループとして管理でき、メンバーの入れ替えが可能です。
- Siteライセンス
- 特定の研究サイト内のどのコンピュータに何台でもインストールできます。
機能による分類
利用可能な機能に応じて5種類のライセンス形態が存在します。詳細は、技術情報の機能比較表をご参照ください。
- Essential版
- 従来の機能。
- Advanced版
- ドッキング機能、アポサイト・モデリング、MDトラジェクトリ読み込み機能を付加。
- Advanced with Knime extensions版
- Advanced版+KNIME拡張に対応。
- Expert版
- MDトラジェクトリ・ファーマコフォア解析・MDトラジェクトリ・ファーマコフォアグラフ機能を付加。KNIME拡張に対応。
- Knime extensions版
- KNIME拡張に対応+IL ファーマコフォアデータベース。
評価ライセンス
無償評価ライセンスをご提供可能です。
ご希望の場合、こちらの評価ライセンス申請フォームからご申請いただくか、弊社営業までお問い合わせください。
価格情報
LigandScoutのライセンス価格は、所属機関(一般・官公庁・教育機関)とライセンス(1-seat/Group/Site)の形態、利用可能な機能によって異なります。ライセンス形態と価格については、お問い合わせフォームからお問い合わせいただくか、弊社営業までお問い合わせください。
一般用
| LigandScout 4 Essential 1-year |
1-seat/Group/Site |
|---|---|
| LigandScout 4 Advanced 1-year |
1-seat/Group/Site |
| LigandScout 4 Advanced with Knime extensions 1-year |
1-seat/Group/Site |
| LigandScout 4 Expert 1-year |
1-seat/Group/Site |
| LigandScout Knime extensions 1-year |
1-seat/Group/Site |
官公庁用
| LigandScout 4 Essential 1-year |
1-seat/Site |
|---|---|
| LigandScout 4 Advanced 1-year |
1-seat/Site |
| LigandScout 4 Advanced with Knime extensions 1-year |
1-seat/Site |
| LigandScout 4 Expert 1-year |
1-seat/Site |
| LigandScout Knime extensions 1-year |
1-seat/Site |
教育機関用
| LigandScout 4 Essential 1-year |
1-seat/Site |
|---|---|
| LigandScout 4 Advanced 1-year |
1-seat/Site |
| LigandScout 4 Advanced with Knime extensions 1-year |
1-seat/Site |
| LigandScout 4 Expert 1-year |
1-seat/Site |
| LigandScout Knime extensions 1-year |
1-seat/Site |
FAQ
FAQはこちら
最終更新日:2024/05/13
