PeakTrace
サンガー法によるDNAシーケンシス・トレースの読み取り品質と配列長を改善するために新しく開発されたベースコーラ―およびトレース・プロセッサ
製品概要
「PeakTrace Basecaller™」は、サンガー法によるDNAシーケンスの読み取りの品質と塩基配列長を改善するために新たに開発されたベースコーラー(Basecaller)およびトレース・プロセッサです。また、ABI310,3700,3130,3730,3500を含む、ABI社の全機器から得られたシークエンス・データに対応します。PeakTrace BasecallerはABI KB Basecallerと比較して高品質な塩基数が50%も飛躍したほか、現在流通しているどんなベースコーラーよりも波形の改善がなされており、かつエラー率も低減されています。
小~中規模の施設向けシステム「PeakTrace RP™」は実際の計算をPeakTrace server上でリモートで行い、KBやTraceTunerやphred等の従来のサンガー法によるDNAシーケンシス解析用のBasecallerよりも、質の高いBasecallingを提供します。使用頻度が低いエンドユーザは、「PeakTrace RP™」と同様に、Technelysium社とコラボレーションした「PeakTrace for Chromas」を使用することが可能です。
中~大規模の施設向けに設計された独立型のハードウェアシステム「PeakTrace:Box™」は、グラフィカル・ユーザー・インターフェイスで使用できるほか、コマンドライン・バージョンでLinuxベースの既存LIMSインフラに組み合わせることもできます。ユーザーは、アウトプット・ファイル・フォーマットやトレース・エンハンスメント、トリミング・セッティングといった様々なbasecallingやトレース・プロセッシングのパラメータを、完全にコントロールすることができます。「PeakTrace:Box™」は、ABI、SCF両方の拡張子でトレース・ファイルを作成できるほか、SEQ(FASTAあるいはABI)やQUAL,PHD.1、QualTRACE Ⅲ TraceReportのように様々なテキスト・ファイルも作成することができます。
開発元:オーストラリア Nucleics Pty Ltd
技術情報
バリデーション
目的
現在の改良されたBasecallersを用いると、簡単な方法で読み取る配列の長さを拡大することができます。しかしながら、新しいBasecallersを商品として販売する前には、そのBasecallersの予測される品質スコアと実際に得られる品質スコアが一致しているかバリデーションする必要があります。このバリデーション(品質スコアマッピングとして知られる)により、その新しいBasecallerから出力されたデータは正確であり、発生する問題はBasecallersのプログラム設定によるものではないことを保証します。
Peak Traceを用いると、簡単に読み取る配列の長さを伸ばすことができます。ここでは、ABI社のKBとPeak Traceを用いバリデーション(品質スコアマッピングを用いて)を行うことで、Peak Traceで出力されたデータが正確であることを示します。
方法
本研究では、2つの175kBの鮭のBACクローン由来のpUCサブクローンより回収した2203塩基配列を用いて、ABI社のKBvl.2とNucleics社のPeakTraceを比較しました。
最初に間違った塩基配列や混合した配列を除去するために、Nucleics社のQualTraceQCを用いて、その2203塩基配列のデータを解析しました。
続いて、確実に類似した塩基配列を比較できるように、PeakTraceとKBの両ソフトウェアで読み取れる配列を含むようにして解析を行いました(その配列にはPeakTraceで解析すると10個以上のQ20 +塩基が付加されます)。
次は、BLAST(NCBI)を用いて、それぞれのBACクローンのコンセンサス配列と解析した塩基配列を並べて比較しました。BACクローンの塩基配列を並べて比較しました。BACクローンの塩基配列と一致しなかった塩基配列についてはこれ以上解析ができないので排除しました(一致しなかった配列のほとんどは大腸菌K12のゲノムDNAの汚染によるものでした)。
以上の4つのスクリーニング基準を満たした塩基配列数は合計1643でした。そして、BLASTにより並べたその塩基配列を用いて、EwingとGreenの研究内容(参考文献1)を参考に、前1643塩基配列数または観測されたエラーを計算しました。つまり、各アライン塩基に対して、正確・不正確なBasecallの合計を記録するようにしました(観測された品質スコアあるいはQ)。アライン塩基の合計および各Basecaller品質スコア予測の精度も測定するため、それらを各Basecallerの予測されるエラー率(予測Qスコア)と比較しました(Qスコアマッピングとして実行)。
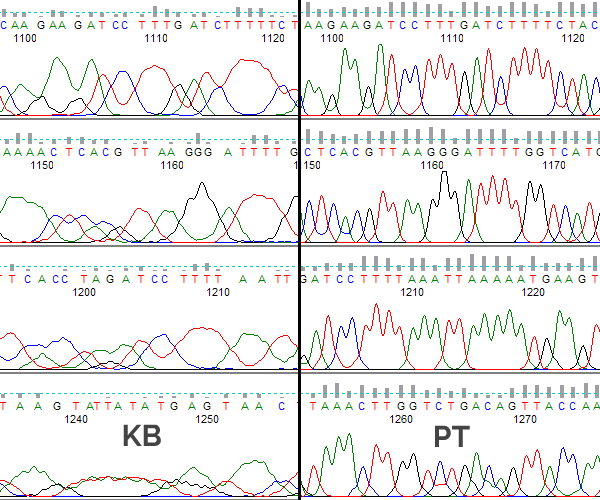
図1. KBとPeakTrace(PT)ベースコーラ―の比較結果(同一トレースファイル使用)
図2. KBベースコーラ―によるBLASTアライメント; 1%エラーリード(解読)長:1275塩基
図3. PeatTraceベースコーラ―によるBLASTアライメント; 1%エラーリード(解読)長:1486塩基
結果
Qスコアマッピングの結果を図4に示しました。結果は、KBよりPeakTraceのほうが正確に実際のエラー率を予測するということが分かりました。特にこの精度の向上は、KBで予測されていた塩基配列の品質を大きく上回るQ20からQ30までの中間品質スコアで非常に顕著です。
表1. KBとPeakTraceベースコーラ―のBLASTアライメント比較結果(図2,3)
| KB | PeakTrace | |
|---|---|---|
| アライメントされた解読塩基数 | 1379 | 1540 |
| 最初のエラーがある解読塩基 | 1084 | 1251 |
| 1%エラー解読塩基数 | 1275 | 1486 |
図4. KBとPeakTraceの予測品質の比較(Observed vs Predicted)
| KB | PeakTrace | |
|---|---|---|
| Q20+塩基数 | 796 | 955 |
| アラインされた塩基数 | 868 | 1031 |
表2. KBとPeakTraceで解析し、BLASTを用いてアラインした平均塩基配列数
結論
PeakTraceで読み取る塩基配列の長さは19%増加し、Q20+の塩基配列については20%の増加が見られました(表2)。本研究での各Basecallerのデータ集積は、塩基配列1000番目から1050番目までの塩基配列間でKBの最適化による解析が停止したので、PeakTraceを使用する際は、既定の実行モジュール時間を2~3分延長してからデータを収集することをお勧めします(すなわち塩基配列1200番目から1300番目の間に解析を中止するということです)。このようなセッティングで解析される結果を見ると、PeakTraceのパフォーマンスがKBと比べ大いに向上していることが分かります。
参考文献
1.Ewing B. & Green P.(1998). Base-calling of automated sequencer traces using phred.Ⅱ.Error probabilities. Genome Res.8(3):186-194
バージョン情報
PeakTrace 6.961(2023年6月14日リリース)
- アーカイブ機能に関するマイナーバグフィックス(コマンドライン版に影響はありません)
PeakTrace 6.96(2023年5月22日リリース)
- (新機能)polyファイルの出力をサポートしました。
- (新機能)以下の実行条件と計測器に対応しました。
- ABI 310
- 50cm POP7 BigDye 3
- 36cm POP4 Rapid BigDye 3
- 30cm POP4 Rapid BigDye 3
- ABI 3200 (SeqStudio)
- 12kV POP1 BigDye 3
- 9kV POP1 BigDye 3
- 4kV POP1 BigDye 3
- ABI 3500
- 50cm POP6 BigDye 3
- 50cm POP6 BigDye 1
- ABI 3600 (SeqStudio Flex)
- RapidSeq50 POP7 BigDye 3
- FastSeq50 POP7 BigDye 3
- ABI 310
- トレースファイルの出力フォーマットとして.scfを選択した際、ログファイルが.ab1の拡張子ではなく.scfがついたファイル名を表示するようになりました。
- バグフィックスとコードの改良
PeakTrace 6.952(2022年10月10日リリース)
- 軽微なバグを修正しました。
PeakTrace 6.951(2020年9月9日リリース)
- 軽微なバグを修正しました。
PeakTrace 6.95(2020年9月1日リリース)
- (新機能)-ksオプションを使用して、処理されたトレースにバブルスパイクを保持します。
- (新機能)Auto PeakTrace 6 は、オプションウィンドウを終了する際に、手動で保存せずに変更を保存するかどうかをチェックします。
- 混合ベースコールが改善されました。
- 稀に起こるトレースタイプのクロストーク補正が改善されました。
- バブルスパイクのあるトレースの処理とベースコールが改善されました。
- 早期ベースコールを改善しました。
- バグを修正しました。
PeakTrace 6.94 (2019年12月21日リリース)
- (新機能)ABI377とABI310の.ab1ファイルが品質スコア付きの.ab1形式で出力されるようになりました。
- (新機能)強制処理の設定で、ベースコールや処理済みのトレースデータがないトレースファイルを.ab1形式で出力できるようになりました。
- (新機能)以下の実行条件と計測器に対応しました。
- ABI310
- 30cm POP4 BigDye 3 & 1
- 30cm POP6 BigDye 3 & 1
- 36cm POP4 BigDye 1
- 50cm POP6 BigDye 3 & 1 Rapid Run
- ABI3700
- POP6 BigDye 3 & 1
- POP5 BigDye 3 o ABI3130/3730
- 50cm POP7 ET terminator
- 50cm POP6 ET terminator
- ABI3130
- 80cm POP4 BigDye 3 & 1
- ABI3200
- 9kV BigDye 1
- 4kV BigDye 1
- MJResearch Biostation
- ABI310
- 40 ベースのウィンドウで q 平均トリムのデフォルト値が 9 に変更されました。
- 良好なベース改善のデフォルト値が -10 に変更されました。
- エラーコード 25 の説明が “正規化に失敗しました” から “過負荷トレース” に変更され、この結果の原因がより明確になりました。
- 混合ベースコールを改善しました。
- Dye Blob “領域でのベースコールを改善しました。
- PCR産物トレースの処理を改善しました。
- バグを修正しました。
動作環境
PeakTrace RP™ 最小構成
バージョン6.951 (2020年9月15日現在)
- Windows XP/Vista/7/8/10(*1) または MacOS X 10.6以降
- インターネット接続環境
- RAM 1GB
- 10GB 空きディスク容量
- 1024×768 以上の画面解像度
PeakTrace:Box™ 最小構成
バージョン6.951 (2020年9月15日現在)
- グラフィカル・ユーザー・インターフェイス
- Windows XP/Vista/7/8/10(*2)
- SSE3をサポートするIntel Core CPU ※CPU性能が律速になるため、最新のCPU使用を推奨します
- RAM 2 GB
- 25 GB 空きディスク容量
- 1024×768 以上の画面解像度
- PeakTrace USB Key with a valid license and firmware 2.04 以降
- CodeMeter Runtime 6.70a 以降
- コマンドライン・バージョン
- 64-bit Linux (REHL7/CentOS7以上)
- 専用ライセンス(*3)
- 専用シリアルナンバー(*4)
※仮想化環境(VMware, Xen等)では動作しませんのでご注意ください。
(*1) Windows Server 2003, 2008, 2012でもおそらく動作しますが、十分なテストは行っておりません。
(*2) Windows Server 2003, 2008 は正式サポートOSではありませんが、ご希望があればお問合せください。
(*3) PeakTrace 5.92 以前のバージョンをお使いの場合は、開発元 Nucleics Pty Ltd あるいは弊社営業担当までお問合せください。
(*4) PeakTrace 6.11 以前のバージョンをお使いの場合は、開発元 Nucleics Pty Ltd あるいは弊社営業担当までお問合せください。
ライセンス形態
ラインナップ
PeakTrace RP™ / PeakTrace Chromas
- PeakTrace RP™
- 小~中規模の施設向けのPeakTraceで、ユーザー・インストール・バージョンです。データ処理はNucleics社PeakTraceサーバーで行われます。
- PeakTrace Chromas
- トレースビューアとベースコーラーが組み合わされた、小規模施設またはエンドユーザー向けに設計されたものです。PeakTraceの簡易利用バージョンです。
PeakTrace:Box™
- 中~大規模の施設向けに設計された、独立型のハードウェアシステムです。高度なデータセキュリティでもオフラインで稼働できるシステムです。PeakTrace:Box 1ライセンスに付き、1つのUSBライセンスキーが付属します。
評価ライセンス
無償評価ライセンスをご提供可能です。
ご希望の場合、こちらの評価ライセンス申請用フォームからご申請いただくか、弊社営業までお問い合わせください。
価格情報
PeakTrace Basecaller™のライセンス価格は、製品の形態、units数によって異なります。ライセンス形態と価格については、お問い合わせフォームからお問い合わせいただくか、弊社営業までお問い合わせください。
PeakTrace RP™ / PeakTrace Chromas
| 商品名 (units数) | 価格(税別) |
|---|---|
| 100 PeakTrace RP units | お問合せください |
| 1000 PeakTrace RP units | お問合せください |
| 2000 PeakTrace RP units | お問合せください |
| 5000 PeakTrace RP units | お問合せください |
| 10 000 PeakTrace RP units | お問合せください |
| 20 000 PeakTrace RP units | お問合せください |
| 50 000 PeakTrace RP units | お問合せください |
※上記価格は予告なく変更される場合があります。
PeakTrace:Box™
| 商品名 (Traces per Year) | 価格(税別) |
|---|---|
| up to 10 000 units for 24 month | お問合せください |
| up to 30 000 units for 24 month | お問合せください |
| up to 100 000 units for 24 month | お問合せください |
| up to 300 0000 units for 24 month | お問合せください |
| up to 3 000 000 units for 24 month | お問合せください |
FAQ
FAQはこちら
最終更新日:2023/08/03